上海吉凯基因医学科技股份有限公司品牌商
15 年
手机商铺
- NaN
- 0
- 0
- 1
- 0
公司新闻/正文
想做特异性表达,先get“启动子预测”技能!
7024 人阅读发布时间:2019-03-15 10:21
近期与科研工作者沟通过程中,经常被问到:
“xxx启动子的序列帮我查询一下。”
“有好用的软件推荐一下吗?”
“帮我看看我预测的启动子是否合适?”
“为什么我预测的启动子序列和技术支持提供的不一致?”
“怎么说我预测的序列含有‘启动子位于编码基因翻译起始位点上游序列’的错误信息?”
“能不能来一篇启动子预测的文章呀?”
。。。。。。
看到大家有这么多的问题,又对启动子预测如此感兴趣!小编甚是开心。
今天就给大家介绍一下“常规基因的启动子预测方式”(适用于lncRNA,mRNA)。
启动子是位于结构基因5'端上游的DNA序列,能活化RNA聚合酶,使之与模板DNA准确的结合并具有转录起始的特性。常规启动子以二型为主,核心序列包含TATA box和CAAT box,这两部分组成的序列含有基础转录活性,也就是具备启动子特征,但是表达水平很低,此序列紧挨着转录起始位点TSS,一般位于编码基因5UTR(不是ATG哦)上游的300bp之内。
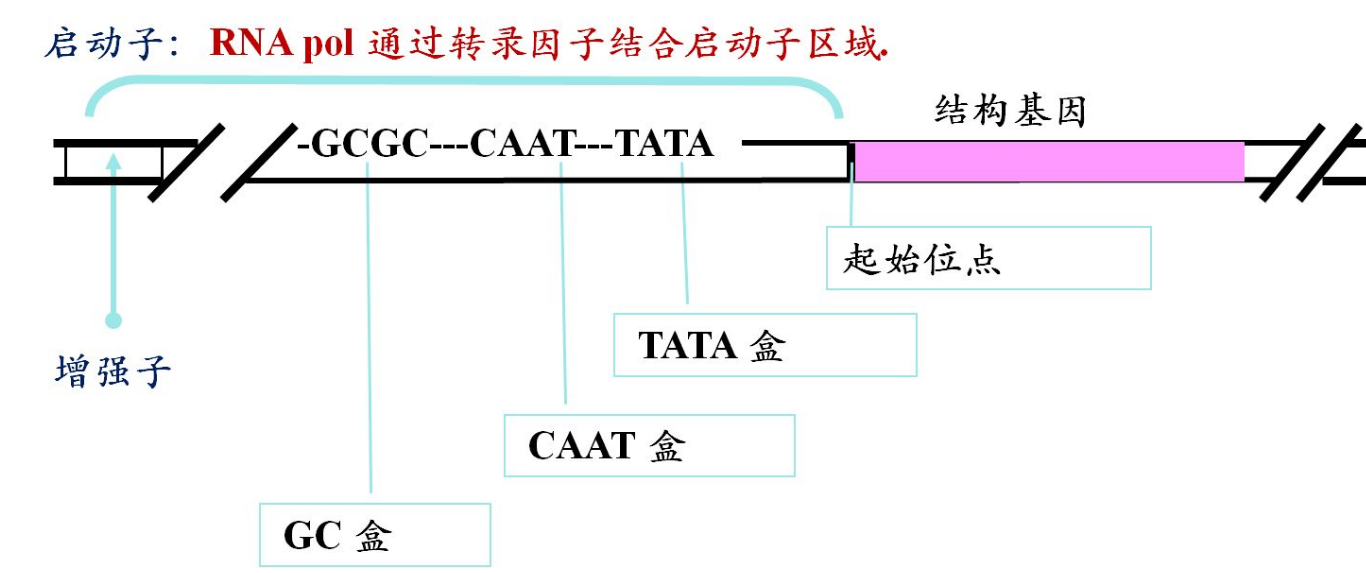
研究启动子,其实不是研究核心启动子,而是研究启动子在细胞不同状态下表达活性的差异性,这种差异由启动子临近基序调控,研究较多的是增强子、甲基化、转录因子,这些基序位于核心启动子上游居多,所以,常规取包含核心启动子及其上游共计2kb进行研究。
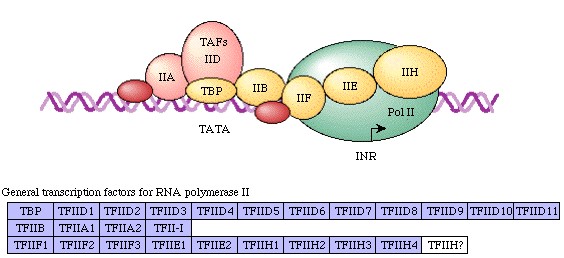
转录因子-启动子结合图

打开NCBI主页GENE链接,在搜索框输入对应基因及物种(https://www.ncbi.nlm.nih.gov/gene/ )
打开搜索的结果,进入基因界面,小编划重点了哈~

注意基因位于染色体正义链还是反义链—看箭头指向,如果是正义链,则启动子取Location左侧数字减去2000bp;如果是反义链(染色体写法会加上complement),则取Location右侧数字加2000bp。
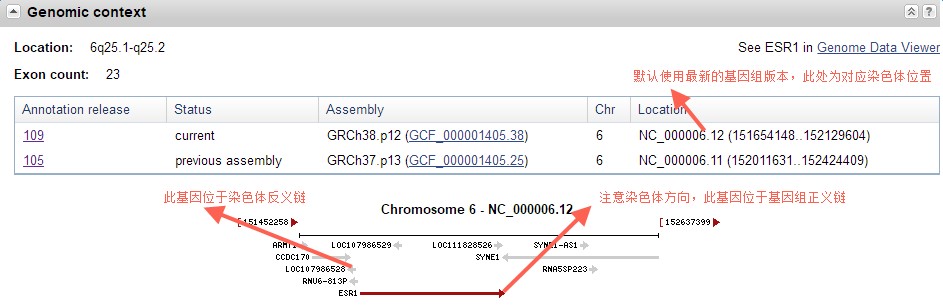

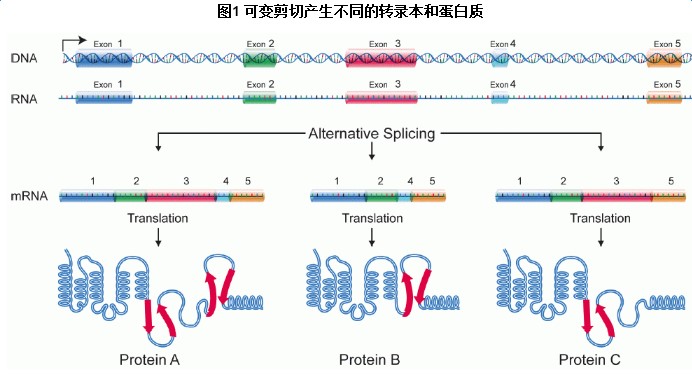
根据转录本确定启动子实际所在位置。很多科研工作者认为基因组延伸的序列就是启动子,或者一个基因只有一个启动子,实际上这是错误的。基因通过选择性剪切,从RNA中选择外显子的不同部分以形成不同的mRNA序列,每个独特的mRNA序列产生独特的蛋白质,这些高度相似的蛋白质亚型可以由可变剪切、可变启动子或转录后修饰形成。
所以,研究基因得确定候选的转录本亚型,转录本亚型可从上述基因界面获得,该界面对不同亚型会有具体分类(如isoform 1;isoform a等),亚型不同,启动子可能会有差异,可在Genomic regions, transcripts, and products处查看:
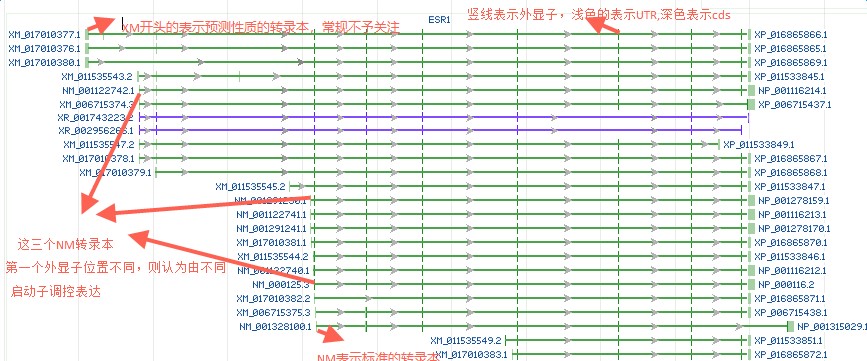
ESR1转录本较多,如果该基因默认取延伸的2kb,那么实际上取的是最头部的转录本上游序列,所以对应的是XM_017010377的启动子,显然不符合常规的研究,那么该如何操作呢??
小编接着往下细说:

首先,您得有个序列分析软件,推荐snapgene,
可在https://www.snapgene.com/release-notes/?referrer=SnapGene%20Viewer 下载

打开研究的转录本亚型,如NM_000125,点击软件Import,输入NM_000125,点击Import,打开序列,复制第一个外显子
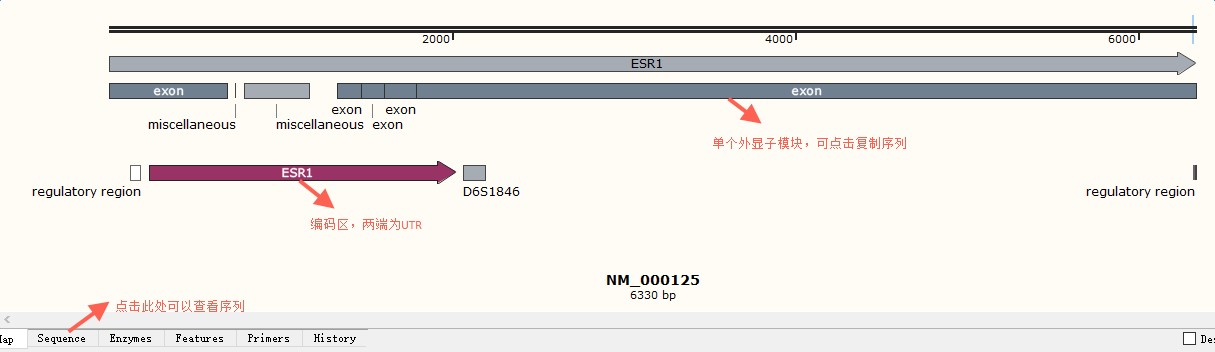

查找基因组序列:
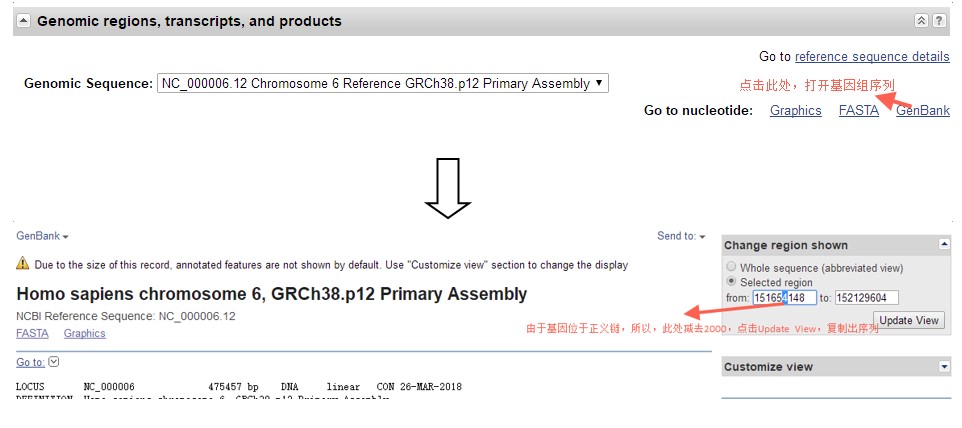
Copy出全长序列以snapgene打开,并以第二步复制的第一个外显子在基因组序列搜索,其上序列就是启动子座落区,复制出来即可。
如果担心取的启动子有误怎么办?
不要担心,从EPD数据库 (https://epd.epfl.ch//documents.php)查找比对,EPD收录的是已经验证的真核生物二型启动子数据库,以基因名称+物种搜索:
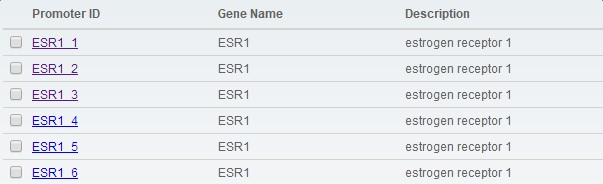
搜人源ESR1获得收录的6个启动子,我们看下前三个启动子位置:
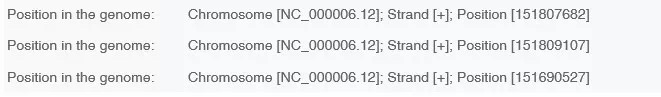
由启动子位置看出,不同转录本的启动子位置差异较大。接下来可以从Get sequence获得序列,然后跟前期取的启动子比对进行确认。
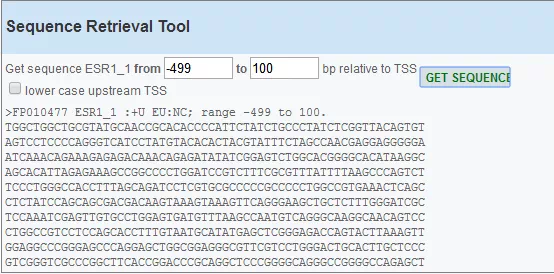
该网站还提供JASPAR数据库来源的转录因子—启动子结合预测,选择对应参数即可:
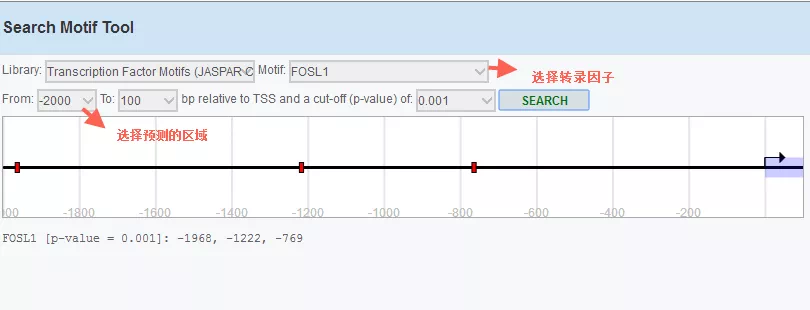
以上是常规启动子获取的方式,是不是很easy呢!!!
吉凯基因将为各位科研工作者提供更多的特异性启动子,为科研助力!!!

活动详情
购买指定腺相关病毒产品目录价满2000元即可享受9折,可与其他优惠活动同享。
截止日期:2019年5月31日
详情请咨询销售工程师或拨打电话:021-51320189-8301