上海吉凯基因医学科技股份有限公司品牌商
15 年
手机商铺
商家活跃:
产品热度:
- NaN
- 0
- 0
- 1
- 0
公司新闻/正文
干货| 增强子SNP预测、实验操作指南
2833 人阅读发布时间:2020-11-18 13:42
随着分子生物技术的发展,全基因组关联研究(GWAS)已成功鉴定出与复杂疾病或其他表型性状显著相关的众多遗传变异,以单个核苷酸变异为主,简称SNP。有趣的是,许多变异位于基因组的非编码区。这些非编码DNA突变体在分子水平上是否以及如何影响表型性状,在很大程度上仍然未知,那么如何研究呢?小编将用多篇文章分别阐述SNP位于增强子、剪切位点、UTR位置的研究思路,今天谈谈增强子。
SNP位于编码区会直接导致蛋白氨基酸组成发生变化,从而诱发结构或者功能的改变,研究相对容易,只需分别过表达野生型、突变型基因,分析表型;但远离基因座或者位于内含子中的SNP不会对蛋白组成及结构造成变化, 是否有功能呢?往哪个方向研究呢?
我们来看一些案例:
转录因子HLTF、LEF1和MITF通过调节位于色素基因OCA2上游21 kb 的rs12913832,影响OCA2基因表达强度,从而调节人皮肤、眼睛和头发的色素性状[1];
转录因子AP-2α结合IRF6内含子中的rs642961位点,其变异型与唇裂症发生有关[2];
HOTAIR内含子中的rs920778位点与食管鳞状细胞癌有关[3];
通常坐落于增强子内的非编码性SNP,有两种情况:
一、真正的增强子,不依赖于调控蛋白即可影响基因表达,这类研究也比较容易,直接构建包含SNP的一段增强子序列即可;
二、影响转录因子调控,即SNP的不同亚型与转录因子的结合强度不同,造成调控活性的变化,此处的难点为如何预测得到对应的转录因子。
下面小编就以rs10036653(rs+数字为SNP位点的标准命名)位点为例,给大家演示下。

“upstream”意味着位点位于ATG10基因的转录起始位点上游,那么如何预测与之相关的转录因子呢?
一、网站预测
在主页输入框填入“rs10036653”,点击“提交/search”,可见snp的多参数信息,按照下图展开:

Haploreg搜索结果
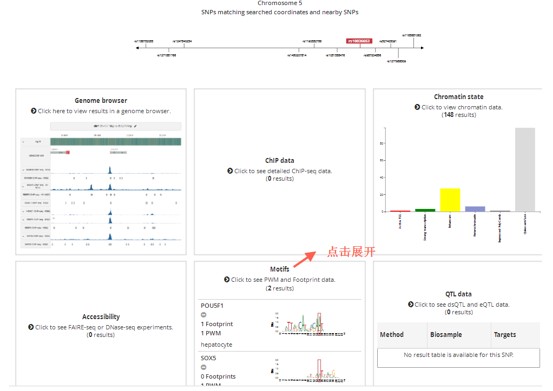
Regulomedb搜索结果
即可得到预测的差异化motifs(motifs:即转录因子结合模型,也就是转录因子):
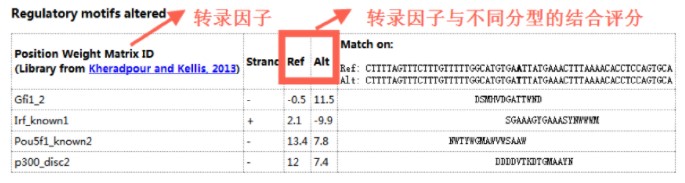
Haploreg预测的差异motifs
Regulomedb预测的差异motifs
通过两个网站分析出与rs10036653相关的转录因子有GFI1、POU5F1、SOX5、IRF1、P300。
二、序列预测
若SNP通过测序得出,无rs编号,或者上述网站未预测到转录因子,可借助转录因子预测网站TFbind分析不同亚型的序列,然后找出差异的转录因子。转录因子与DNA的结合长度一般为5-20bp,因此在snp位点两端各延伸30bp预测即可:
Ref: CTTTTAGTTTCTTTGTTTTTGGCATGTGAATTATGAAACTTTAAAACACCTCCAGTGCA
Alt: CTTTTAGTTTCTTTGTTTTTGGCATGTGATTTATGAAACTTTAAAACACCTCCAGTGCA
输入网站预测(fasta格式):

TFbind输入序列
将预测结果导入excel,删除冗余项(导出数据需分列),然后手动对比评分有差异、或者转录因子ID差异的并且标出,即可得到与snp不同亚型结合的转录因子:
数据整理及对比
这三个方便易用的网站可满足大部分SNP的分析,预测了转录因子,接下来如何做验证呢?
三、CHIP
CHIP:通过染色质免疫共沉淀技术(ChIP),富集与本底或者外源表达转录因子结合的DNA,通过qpcr验证转录因子与不同SNP亚型的结合强度。
多个转录因子与不同SNP亚型的结合强度(CHIP-qpcr)[1]
四、EMSA
EMSA: 凝胶电泳迁移实验,将探针标记DNA片段与细胞裂解液孵育,以电泳迁移率的变化进行定性和定量分析。只需将不同SNP亚型的片段标记探针,与细胞液孵育,以电泳迁移率分析蛋白与片段是否结合。
rs642961亚型与AP-2α的EMSA实验[2]
五、荧光素酶报告实验
研究基因间调控活性,常做的就是荧光素酶报告系统。通过构建报告系统研究SNP位点的增强子与转录因子是否结合最为直接,根据SNP位置不同,不同文章构建方法也是不一样的,具体如下:
1.SNP位于启动子区域(一般5k以内):直接构建启动子全长至PGL3 basic[4]。
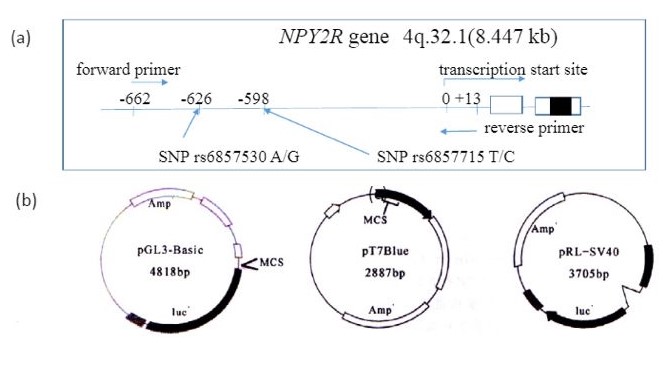
2.SNP位于启动子上游远端:构建SNP位置1k左右的序列与目的基因启动子联合构建至PGL3 basic[2]。
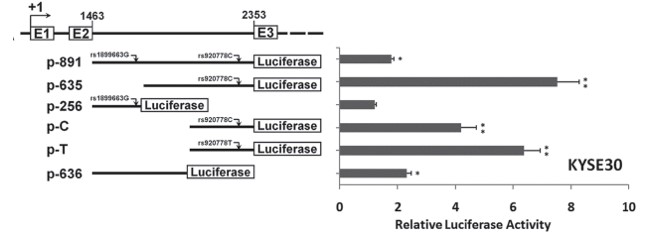
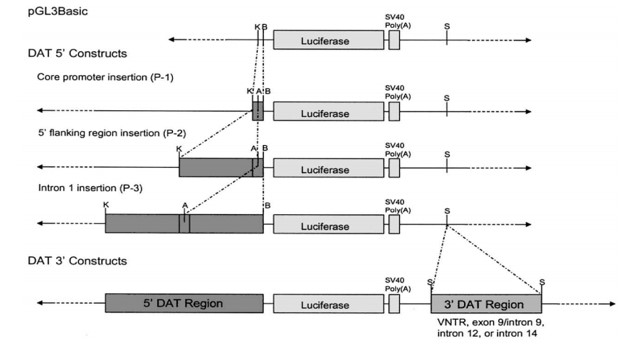
3.SNP位于基因内含子或者基因下游:构建SNP位置1k左右的序列至PGL3 promoter的luc下游,也可将载体本身SV40启动子替换为目的基因启动子[5]。
以上就是增强子SNP的预测及实验操作指南,简单易学。吉凯基因拥有全套荧光素酶报告载体既转录因子过表达现货,可助您快速完成实验所需质粒,快来咨询吧!!!
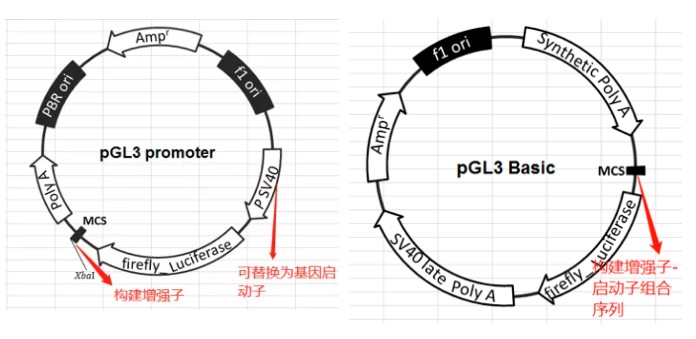
常用的报告载体

扫码添加微信,一站式解决您的科研难题
吉凯基因,年产上万次慢病毒、腺病毒、腺相关病毒;
建有国内大型的慢病毒文库,包含几乎覆盖人类所有基因的,近 15 万个独立克隆;
国内集产品采购和资讯分享的一体化综合服务平台,为科研工作者提供一键式的采购服务,产品周期短至 5 个工作日起;
18 年品质保证、助力客户发表 10000+ 篇 SCI,多项科研成果发表于 Nature Medicine、Nature Immunology、Gastroenterology、Cell Metabolism、Nature Neuroscience 等著名期刊上
建有国内大型的慢病毒文库,包含几乎覆盖人类所有基因的,近 15 万个独立克隆;
国内集产品采购和资讯分享的一体化综合服务平台,为科研工作者提供一键式的采购服务,产品周期短至 5 个工作日起;
18 年品质保证、助力客户发表 10000+ 篇 SCI,多项科研成果发表于 Nature Medicine、Nature Immunology、Gastroenterology、Cell Metabolism、Nature Neuroscience 等著名期刊上
【参考文献】
[1] HERC2 rs12913832 modulates human pigmentation by attenuating chromatin-loop formation between a long-range enhancer and the OCA2 promoter
[2] Disruption of an AP-2α binding site in an IRF6 enhancer is strongly associated with cleft lip
[3] The identification of an ESCC susceptibility SNP rs920778 that regulates the expression of lncRNA HOTAIR via a novel intronic enhancer
[4] Single nucleotide polymorphism- and cell type-dependent gene expression of neuropeptide Y2 receptor
[5] Promoter and intronic variants affect the transcriptional regulation of the human dopamine transporter gene